Kulakovskiy I.V., Favorov A.F., Makeev V.J. (2009) Motif discovery and motif finding from genome-mapped DNase footprint data. Bioinformatics 25(18): 2318-2325.
<up> DMMPMM motif CAD comparison [dmmpmm_compare_html]
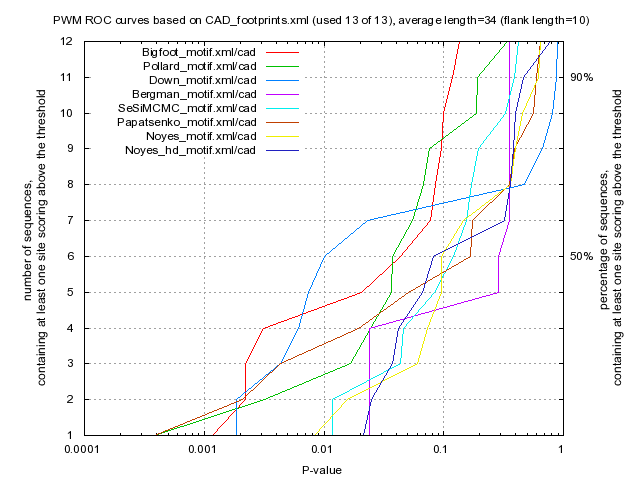 |
motif alignment
motif similarity
|
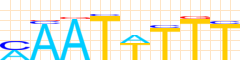
Bigfoot |
Pollard |
Down |
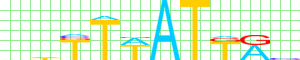
Bergman |
SeSiMCMC |
Papatsenko |
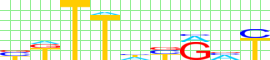
Noyes |
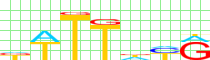
Noyes_hd |
| Bigfoot |
1.0 |
0.2777 |
0.0787 |
0.2229 |
0.1527 |
0.1651 |
0.1131 |
0.109 |
| Pollard |
0.2777 |
1.0 |
0.0424 |
0.1886 |
0.2024 |
0.1851 |
0.0706 |
0.0786 |
| Down |
0.0787 |
0.0424 |
1.0 |
0.1037 |
0.0699 |
0.1083 |
0.0675 |
0.0881 |
| Bergman |
0.2229 |
0.1886 |
0.1037 |
1.0 |
0.1855 |
0.4178 |
0.2836 |
0.2577 |
| SeSiMCMC |
0.1527 |
0.2024 |
0.0699 |
0.1855 |
1.0 |
0.1935 |
0.0763 |
0.0664 |
| Papatsenko |
0.1651 |
0.1851 |
0.1083 |
0.4178 |
0.1935 |
1.0 |
0.1791 |
0.2135 |
| Noyes |
0.1131 |
0.0706 |
0.0675 |
0.2836 |
0.0763 |
0.1791 |
1.0 |
0.6788 |
| Noyes_hd |
0.109 |
0.0786 |
0.0881 |
0.2577 |
0.0664 |
0.2135 |
0.6788 |
1.0 |
|